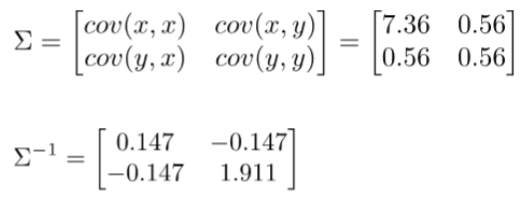TICS-411 Minería de Datos
Clase 13: Detección de Anomalías
¿Qué son las Anomalías?
Definición
- Anomalías
-
Conjunto de puntos que son considerablemente diferentes al resto.
Por definición las Anomalías son relativamente raras. * Pueden ocurrir en proporciones extremadamente bajas en los datos. Ej: 1 entre mil. * El contexto es importante. Ej: Temperaturas bajo cero en Verano.
Ejemplos:
- Telecomunicaciones: Detección de Abusos de Roaming.
- Banca: Compras/Ventas inusualmente elevados.
- Finanzas y Seguros: Detectar y prevenir patrones de gastos fraudulentos.
- Mantención: Predicción de comportamiento irregular/fallas.
- Smart Homes: Detecciones de fugas de Energía
- etc.
Más ejemplos
La definición de Anomalía es altamente subjetiva y depende mucho del Dominio en el cuál se está trabajando.
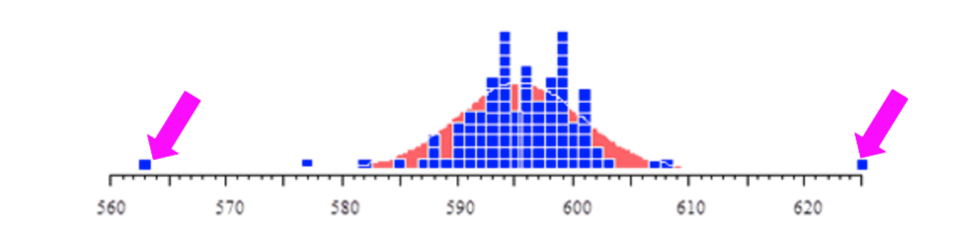
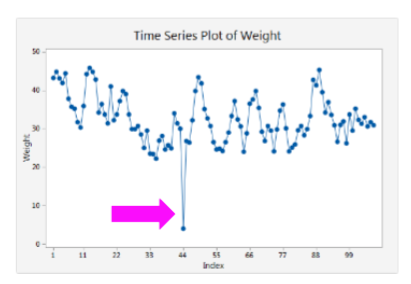
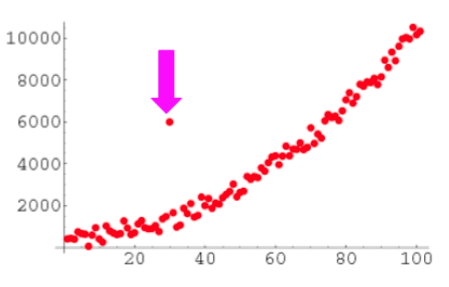
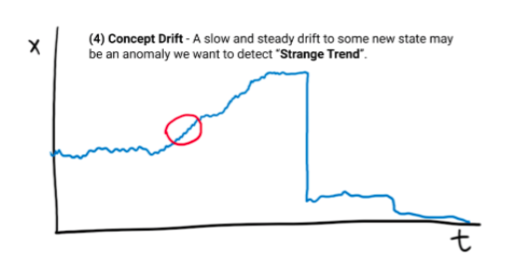
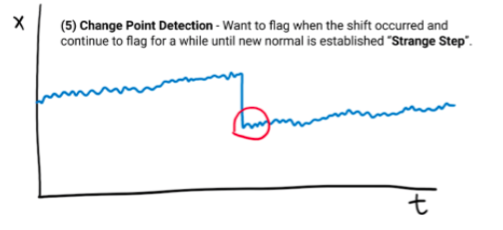
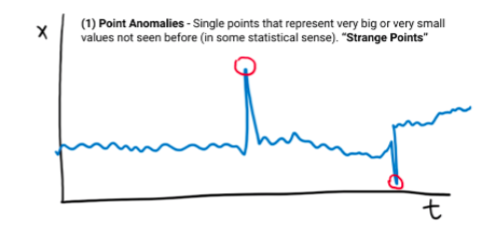
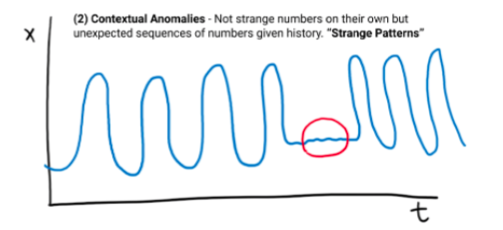
Tipos de Anomalías (Series de Tiempo)
Desafíos
Desafíos
- ¿Cuántos Atributos/Variables usamos para definir un Outlier?
- ¿Cuántos Outliers existen?
- Este tipo de problema suele ser complicado de Etiquetar, por lo que es difícil resolverlo como un problema supervisado.
- Puede ser como “Encontrar una aguja en un pajar”.
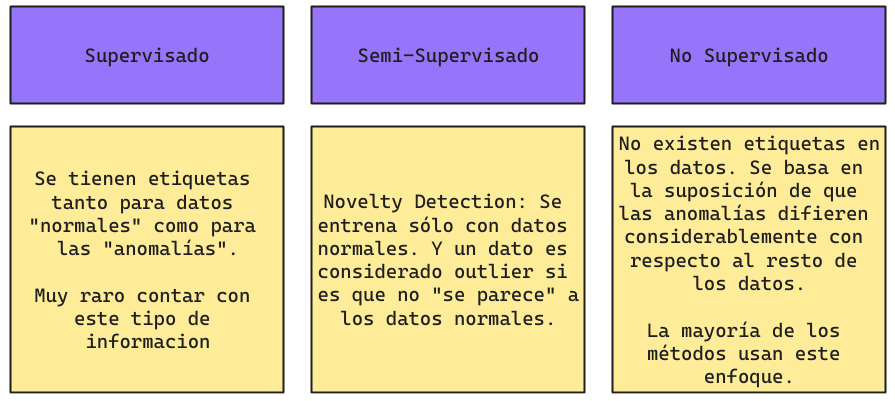
Enfoques
Técnicas de Detección de Outliers
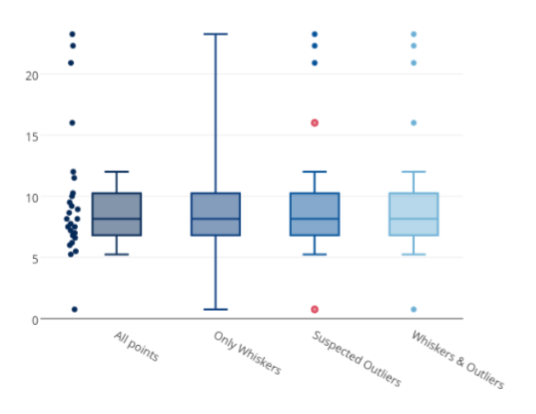
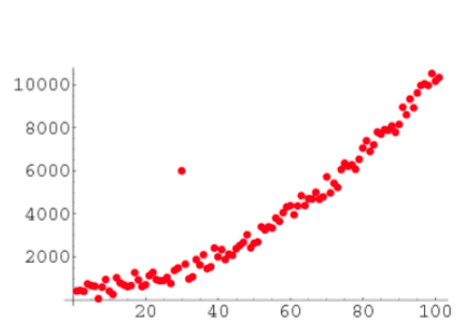
Técnicas Visuales
Estas técnicas son muy subjetivas ya que dependen del criterio/apreciación del usuario.
Técnicas Estadísticas: Test de Grubbs
El test de Grubbs detecta si algún dato es un outlier sobre una variable asumiendo que se distribuyen de manera normal.
\[G = \frac{\underset{i = 1,2,...,n}{max}|x_i - \bar{X}|}{S_x}\]
donde \(\bar{X}\) y \(S_x\) corresponden a la media y Desviación Estándar Muestral.
- Eso implica que \(G\) se distribuye como una t-student de \(n-2\) grados de libertad, por lo tanto si:
\[ G_{critico} = \frac{n-1}{\sqrt{n}}\sqrt{\frac{t^2_{(\alpha/n, n-2)}}{n-2+t_{(\alpha/n, n-2)^2}}}\]
Si \(G > G_{critico}\), \(x_i\) es considerado un outlier con una significancia \(\alpha/n\) para una t-student con \(n-2\) grados de libertad.
Test de Grubs en Python
Este código debiera entregar una lista de todos los puntos que son considerados outliers.
from scipy import stats
import numpy as np
n = 16 # Número de Datos
alpha = 0.05 # nivel de confianza
t_crit = stats.t.ppf(1-alpha/n, n-2)
G_crit = (n-1)/np.sqrt(n)*np.sqrt(t_crit**2/(n-2 + t_crit**2))
data = np.array([5,14,15,15,19,17,16,20,22,8,21,28,11,9,29,40])
G_test = np.abs(data-np.mean(data)/np.std(data))
test_grubbs = np.where(G_test>G_crit)
print(f"Outliers: {data[test_grubbs]}")Caso Multivariado
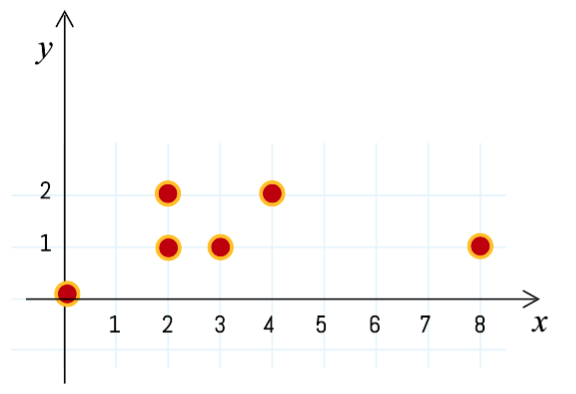
La idea es calcular la distancia de cada punto al centro tomando en consideración la covarianza.
Caso Multivariado
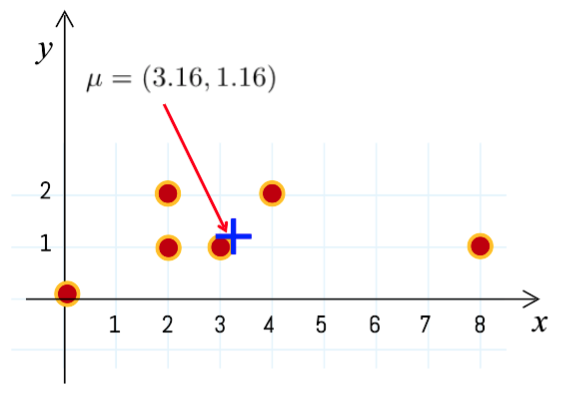
Paso 2
- Calcular la Inversa de la Matriz de Covarianza:
Caso Multivariado: Continuación
Paso 3
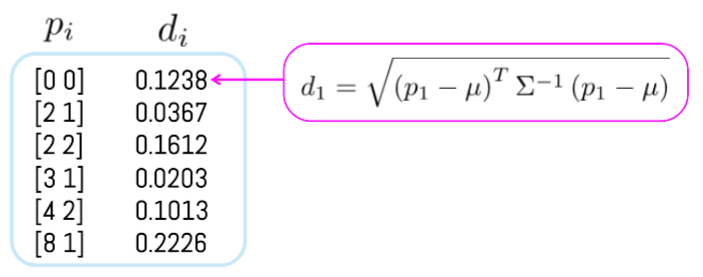
Calcular la distancia de cada punto con respecto a la media y la inversa de la Covarianza.
\[d_i = (p_i - \mu)^T \Sigma^{-1}(p_i - \mu)\]
\[d_1 = (p_1 - \mu)^T \sigma^{-1}(p_1 - \mu)\]
\[d_1 = ([0,0] - [3.16, 3.16])^T \begin{bmatrix} 0.147 & -0.147 \\ -0.147 & 1.911 \\ \end{bmatrix} ([0,0] - [3.16, 3.16])\]
Se debe repetir este procedimiento para cada punto.
Caso Multivariado: Continuación
Paso 4:
Se debe calcular el punto crítico según t-student con 95% confianza, y orden de magnitud \(m\) dimensiones.
\[t_{(\alpha = 0.95,2)} = 5.99\]
Paso 5
Comparar, Si \(d_i>t_{crit}\) entonces \(d_i\) es Outlier.
En este caso ningún valor de \(d_i\) es mayor al $t_{(crit)}, por lo tanto, no hay outliers.
Distancia de Mahalanobis
La distancia de Mahalanobis corresponde a:
\[d_i = \sqrt{(p_i - \mu)^T \Sigma^{-1}(p_i - \mu)}\]
Se puede repetir el mismo procedimiento anterior, sólo que se define una Distancia de Mahalonobis umbral. Las que superen dicho umbral son considerados como Outliers.
Técnicas de Detección basadas en Modelos
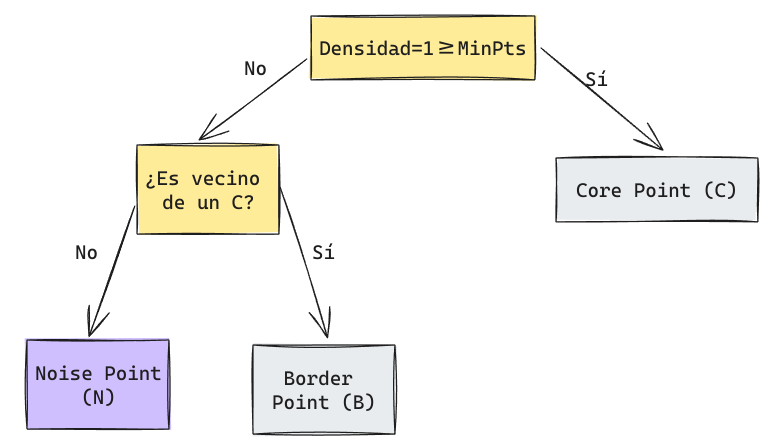
DBSCAN
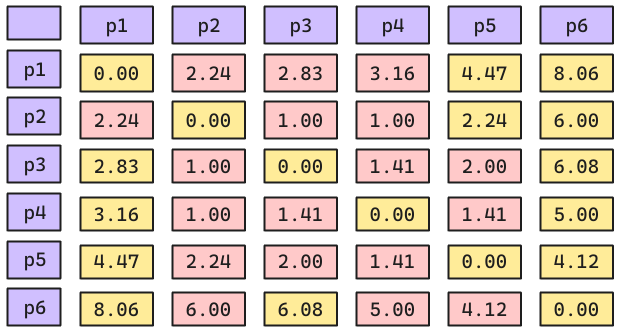
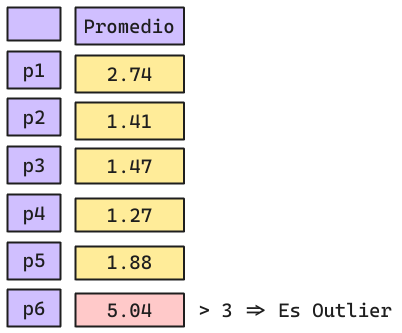
Podemos utilizar el procedimiento que aprendimos de DBSCAN. Todos los puntos Noise serán considerados como Anomalías.
K-Nearest Neighbor
Se puede utilizar los modelos de vecinos más cercanos para determinar outliers siguiendo el siguiente procedimiento:
K-Nearest Neighbor: Continuación
Local Outlier Factor (LOF)
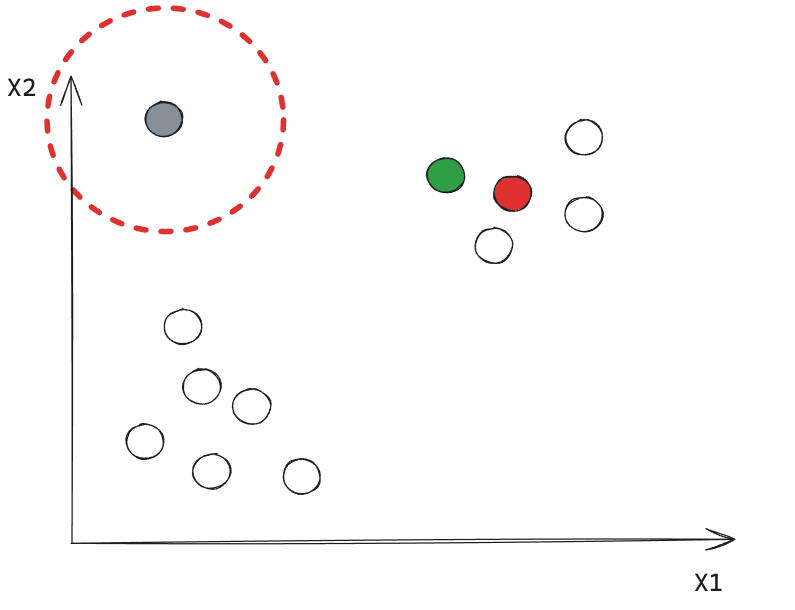
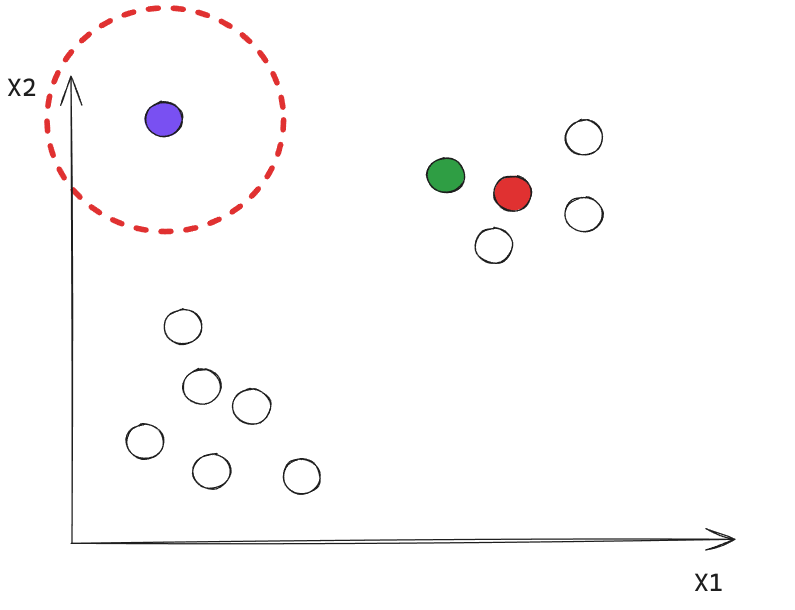
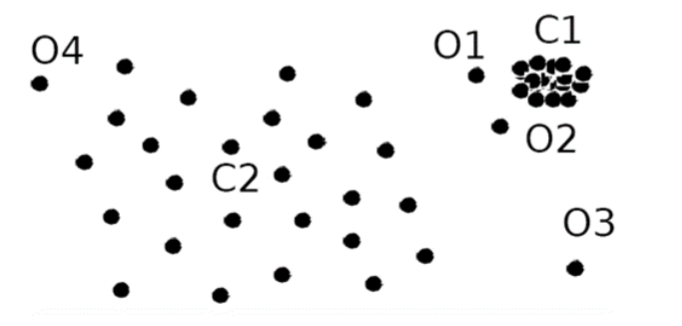
- Local Outlier Factor (LOF) detecta anomalías con sus vecindarios locales, en lugar de la distribución glocal de los datos.
En la Figura, \(O1\) y \(O2\) son anomalías locales en comparación con \(C1\), \(O3\) es una anomalía global, y \(O4\) no es una anomalía.
Algoritmo
- Determinar \(N(x,k)\), los k-vecinos más cercanos de cada punto x.
- Para todo punto \(y\), calcular la distancia a su k-ésimo vecino más cercano.
- Calcular la reach-distance entre todos los puntos: \[reach-distance_k(x,y) = max\{k-distance(y), d(x,y)\}\]
- Calcule la densidad del vecindario local sobre sus \(k\) vecinos, donde \(|N(x,k)| = k\).
\[density(x,k) = lrd_k(x) = \left(\frac{\sum_{y \in N(x,k)} reach-distance_k(x,y)}{|N(x,k)|}\right)^{-1}\]
- Calcule el
Local Outlier Factorpara el punto x como la proporción de la densidad de sus \(k\) vecinos más cercanos, con respecto a la densidad del punto \(x\).
\[ LOF(x) = \frac{\sum_{y \in N(x,k) density(y,k)}}{|N(x,k)|density(x,k)} \]
LOF(X) >> 1 implica anomalía.
Ejemplo Local Outlier Factor
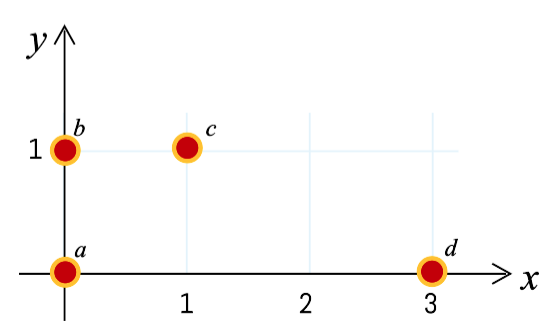
Consideremos los siguientes 4 puntos de datos: a(0,0), b(0,1), c(1,1), d(3,0). Calcular el LOF para cada punto y mostrar la anomalía principal.
Utilizar \(K = 2\) y Distancia Manhattan.
Ejemplo Local Outlier Factor: Continuación
Paso 2: Para todo punto \(y\), calcule la distancia a su k-ésimo vecino más cercano.
- \(dist_2(a) = dist(a,c) = 2\) (c es el 2do vecino más cercano)
- \(dist_2(b) = dist(b,a) = 1\) (a/c es el 2do vecino más cercano)
- \(dist_2(c) = dist(c,a) = 2\) (a es el 2do vecino más cercano)
- \(dist_2(d) = dist(d,a) = 3\) (a/c es el 2do vecino más cercano)
Ejemplo Local Outlier Factor: Continuación
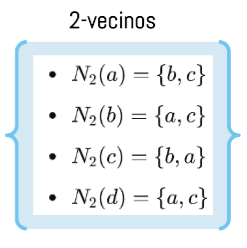
Paso 3: Calcular la reach-distance entre todos los puntos, es decir, los puntos vecindarios a una distancia k.
\(N_k(o)\): Vecindario de \(k\)-distancia de \(o\), \(N_k(o)=\{o'\|o' \in D, dist(o,o') \le dist_k(o)\}\)
- \(N_2(a) = \{b,c\}\)
- \(N_2(b) = \{a,c\}\)
- \(N_2(c) = \{b,a\}\)
- \(N_2(d) = \{a,c\}\)
Ejemplo Local Outlier Factor: Continuación
- \(reach-dist_2(b \leftarrow a) = max\{dist_2(b), dist(b,a)\} = max\{1,1\} = 1\)
- \(reach-dist_2(c \leftarrow a) = max\{dist_2(c), dist(c,a)\} = max\{2,2\} = 2\)
Calcular el resto de manera análoga.
Ejemplo Local Outlier Factor: Continuación
Entonces, \(lrd_k(o)\): Densidad de alcanzabilidad local de \(o\).
\[lrd_2(a) = \frac{|\mathcal{N}(a)|}{reach-dist_2(b\leftarrow a) + reach-dist_2(c \leftarrow a)} = \frac{2}{1 + 2} = 0.667\] \[lrd_2(b) = \frac{|\mathcal{N}(b)|}{reach-dist_2(a\leftarrow b) + reach-dist_2(b \leftarrow b)} = \frac{2}{2 + 2} = 0.5\] \[lrd_2(c) = \frac{|\mathcal{N}(c)|}{reach-dist_2(b\leftarrow c) + reach-dist_2(a \leftarrow c)} = \frac{2}{1 + 2} = 0.667\] \[lrd_2(d) = \frac{|\mathcal{N}(d)|}{reach-dist_2(a\leftarrow d) + reach-dist_2(c \leftarrow d)} = \frac{2}{1 + 2} = 0.33\]
Ejemplo Local Outlier Factor: Continuación
Paso 5: Calcular el Local Outlier Factor para el punto \(x\) como la proporción de la densidad de sus \(k\) vecinos más cercanos, con respecto a la densidad del punto \(x\).
\[LOF(x) = \frac{\sum_{y \in N(x,k)} density(y,k)}{|N(x,k)|density(x,k)}\]
\[LOF_2(a) = \frac{lrd_2(b) + lrd_2(c)}{N_2(a) \cdot lrd_2(a)} = \frac{0.5 + 0.667}{2 \cdot 0.667} = 0.87\] \[LOF_2(b) = \frac{lrd_2(a) + lrd_2(c)}{N_2(b) \cdot lrd_2(b)} = \frac{0.667 + 0.667}{2 \cdot 0.5} = 1.334\] \[LOF_2(c) = \frac{lrd_2(b) + lrd_2(a)}{N_2(c) \cdot lrd_2(c)} = \frac{0.5 + 0.667}{2 \cdot 0.667} = 0.87\] \[LOF_2(d) = \frac{lrd_2(a) + lrd_2(c)}{N_2(d) \cdot lrd_2(d)} = \frac{0.667 + 0.667}{2 \cdot 0.33} = 2\]
Ejemplo Local Outlier Factor: Continuación
Paso 6: Ordena todas las LOF_k(o)
- LOF_2(d) = 2 \(\implies\) el punto paraecer una anomalía (LOF >> 1)
- LOF_2(b) = 1.334
- LOF_2(a) = 0.87
- LOF_2(c) = 0.87
Detalles Técnicos
- Dado que esto sigue un enfoque local, la resolución depende de la elección del usuario para \(k\).
- Genera una puntuación (Anomaly Score) para cada punto.
- Como \(LOF\) es una razón, es difícil de interpretar. No existe un valor umbral específico por encima del cual un punto se define como un valor atípico. La identificación de un valor atípico depende del problema y del usuario
- Como \(LOF\) es una razón, es difícil de interpretar. No existe un valor umbral específico por encima del cual un punto se define como un valor atípico. La identificación de un valor atípico depende del problema y del usuario.
ありがとうございました
(Arigato Gozaimasu)