TICS-411 Minería de Datos
Clase 3: Modelación Descriptiva y K-Means
Modelación Descriptiva (Aprendizaje no Supervisado)
Definiciones
- Aprendizaje No supervisado
-
Es un tipo de aprendizaje que no requiere de etiquetas (las respuestas correctas) para poder aprender.
En nuestro caso nos enfocaremos en un caso particular de Modelación Descriptiva llamada Clustering.
- Clustering
-
Consiste en agrupar los datos en un menor número de
entidadesogrupos. A estosgruposse les conoce comoclustersy pueden ser generados de manera global, o modelando las principales características de los datos.
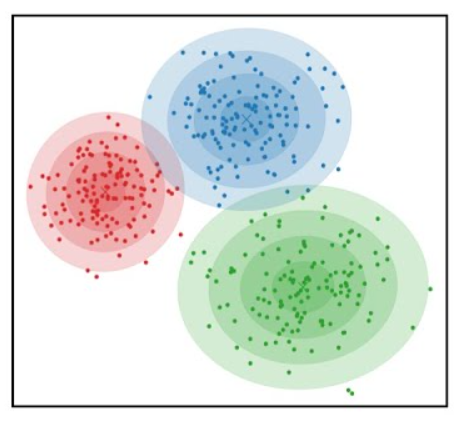
Intuición
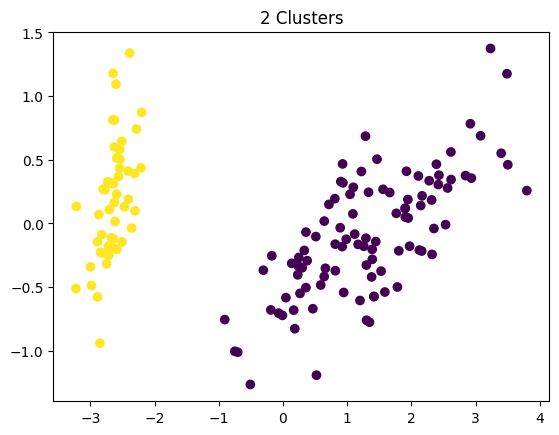
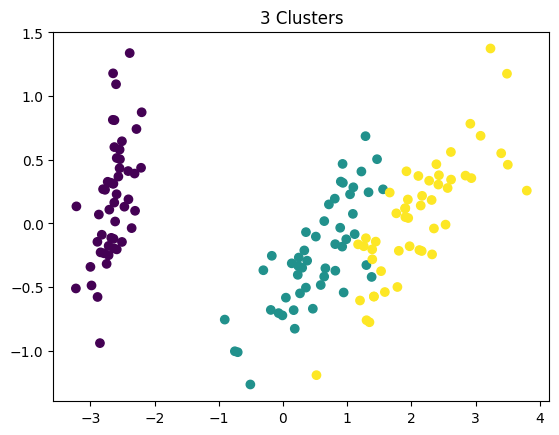
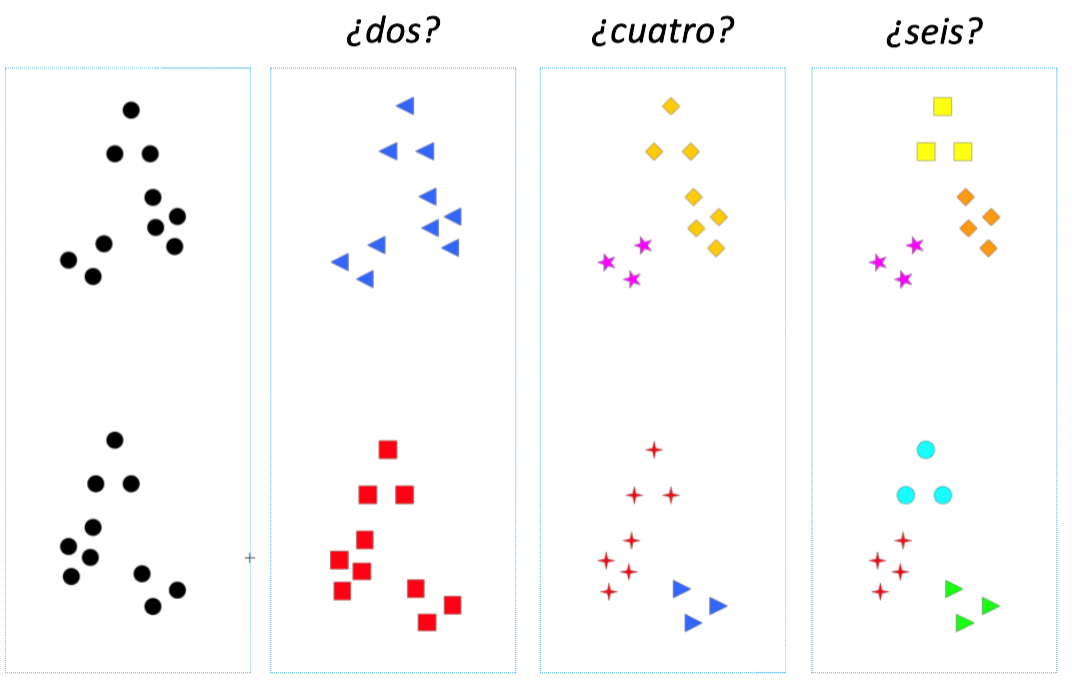
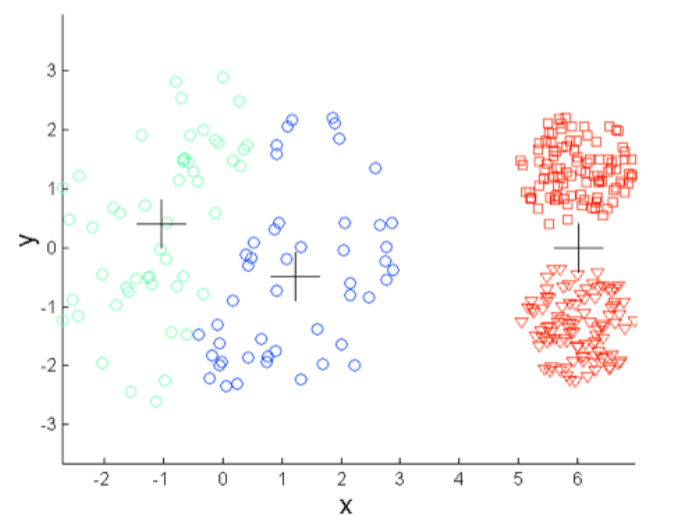
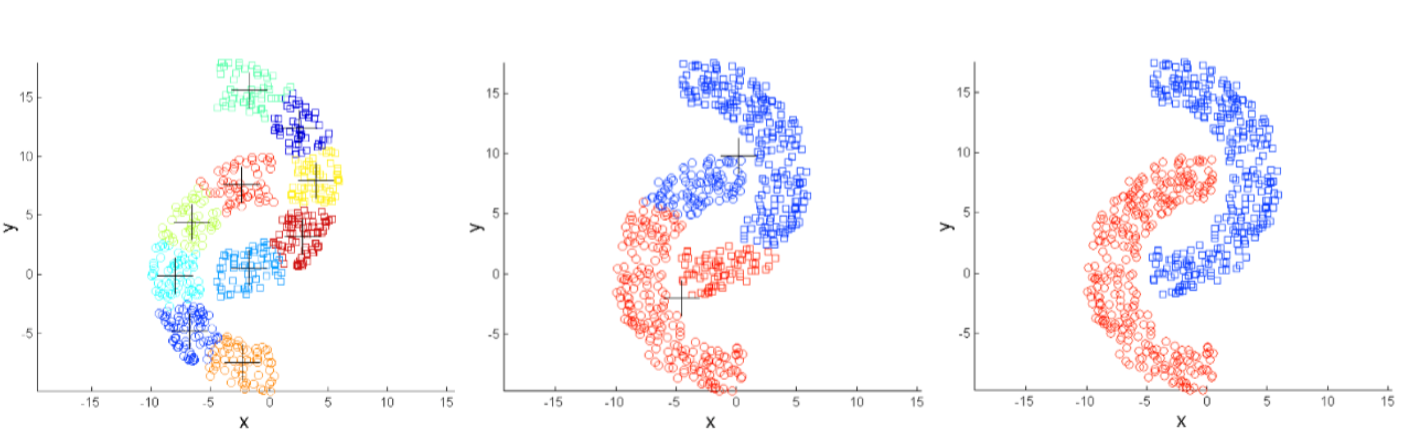
¿Cuántos clusters se pueden apreciar?
Clustering
Clustering: Introducción
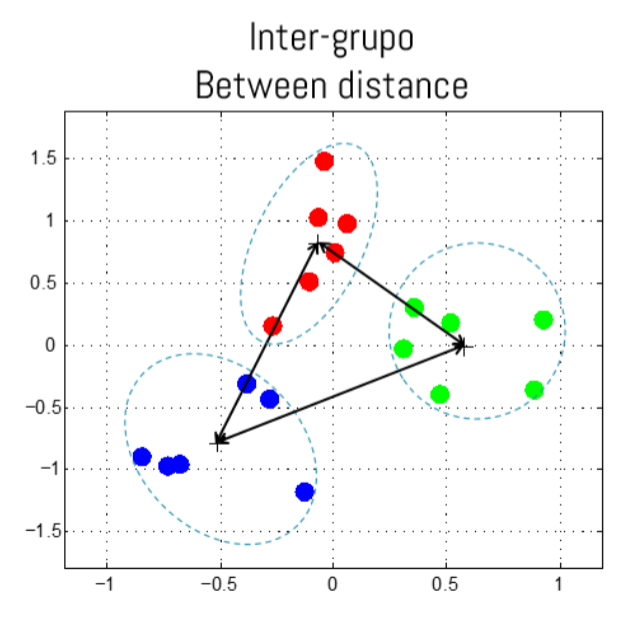
Clustering: Consiste en buscar grupos de objetos tales que la similaridad intra-grupo sea alta, mientras que la similaridad inter-grupos sea baja. Normalmente la distancia es usada para determinar qué tan similares son estos grupos.
Clustering: Evaluación
- Evaluar el nivel del éxito o logro del Clustering es complicado. ¿Por qué?
Clustering: Tipos
Clustering: Partición
Los datos son separados en
Kclusters, donde cada punto pertenece exclusivamente a unúnicocluster.
Clustering: Densidad
Se basan en la idea de continuar el crecimiento de un cluster a medida que la densidad (número de objetos o puntos) en el vecindario sobrepase algún umbral.
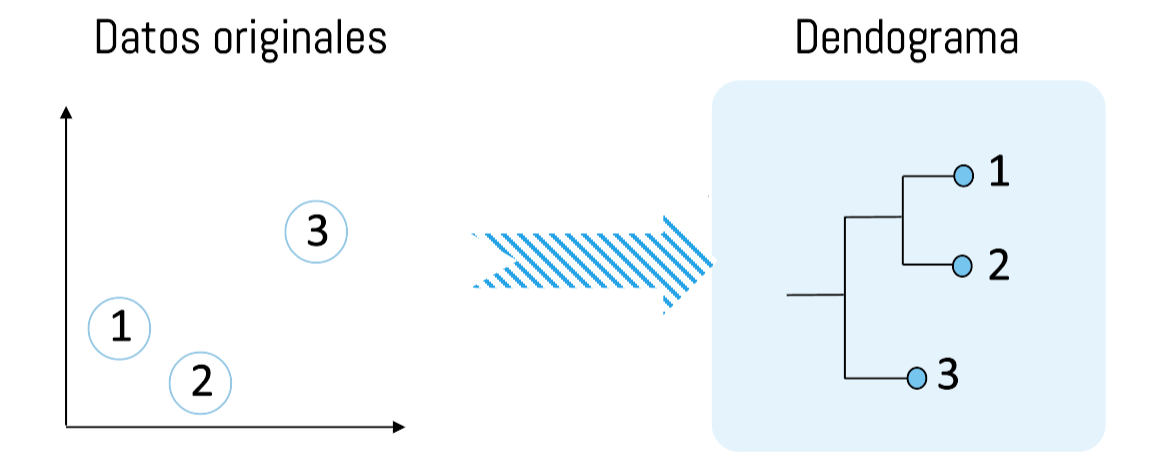
Clustering: Jerarquía
Los algoritmos basados en jerarquía pueden seguir 2 estrategias:
Aglomerativos: Comienzan con cada objeto como un grupo (bottom-up). Estos grupos se van combinando sucesivamente a través de una métrica de similaridad.
Para n objetos se realizan n-1 uniones.Divisionales: Comienzan con un solo gran cluster (bottom-down). Posteriormente este mega-cluster es dividido sucesivamente de acuerdo a una métrica de similaridad.
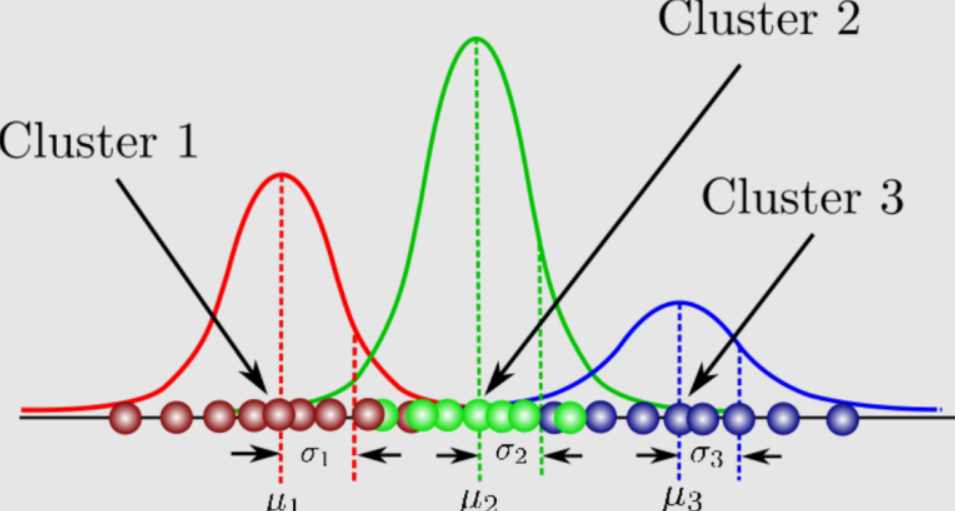
Clustering: Probabilístico
Se ajusta cada punto a una distribución de probabilidades que indica cuál es la probabilidad de pertenencia a dicho cluster.
Métodos Basados en Partición
Partición
Los datos son separados en
KClusters, donde cada punto pertenece exclusivamente a un único cluster. AKse le considera como unhiperparámetro.
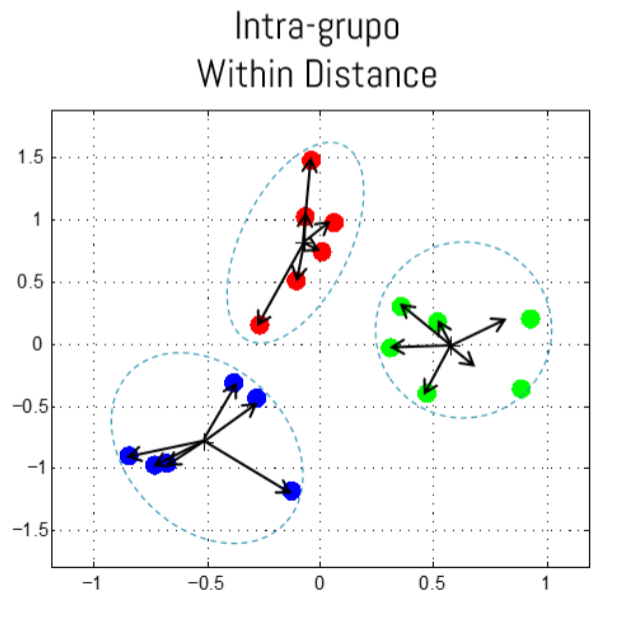
- Cluster Compactos: Minimizar la
distancia intra-cluster(within cluster). - Clusters bien separados: Maximizar la
distancia inter-cluster(between cluster).
\[ Score (C,D) = f(wc(C),bc(C))\]
El puntaje/score mide la calidad del clustering \(C\) para el Dataset \(D\).
Score
\[ Score (C,D) = f(wc(C),bc(C))\]
- Distancia Between-Cluster: \[bc(C) = \sum_{1 \le j \le k \le K} d(r_j, r_k)\]
donde \(r_k\) representa el centro del cluster \(k\): \[r_k = \frac{1}{n_k} \sum_{x_i \in C_k} x_i\]
- Distancia Within-Cluster (Inercia): \[wc(C) = \sum_{k=1}^K \sum_{x_i \in C_k} d(x_i, r_k)\]
Distancia entre los centros de cada cluster.
Distancia entre todos los puntos del cluster y su respectivo centro.
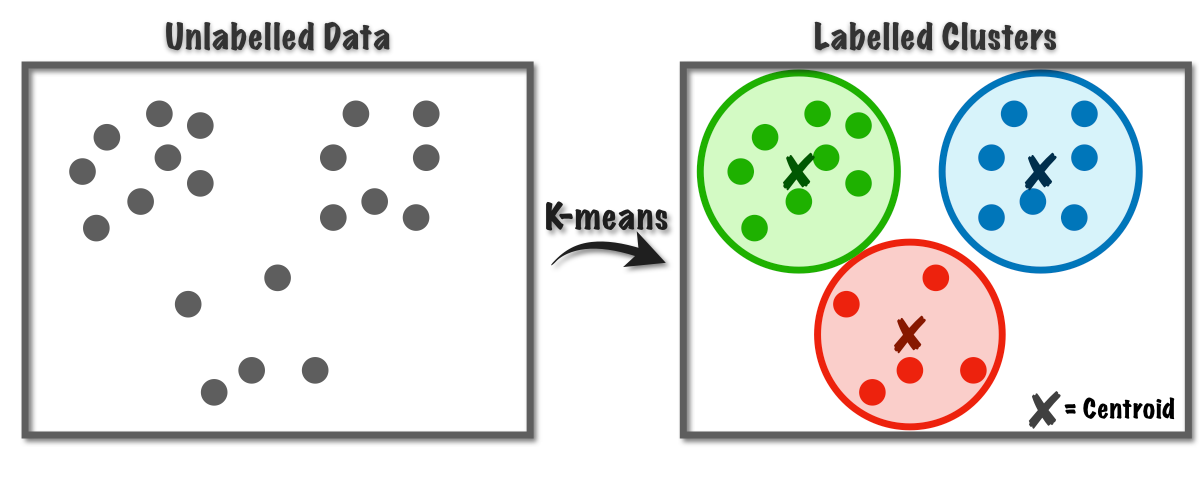
K-Means
- K-Means
-
Dado un número de clusters \(K\) (determinado por el usuario), cada cluster es asociado a un centro (centroide). Luego, cada punto es asociado al cluster con el centroide más cercano.
Normalmente se utiliza la Distancia Euclideana como medida de similaridad.
- Se seleccionan \(K\) puntos como centroides iniciales.
- Repite:
- Forma K clusters asignando todos los puntos al centroide más cercano.
- Recalcula el centroide para cada clase como la media de todos los puntos de dicho cluster.
- Se repite este procedimiento por un número finito de iteraciones o hasta que los centroides no cambien.
K-Means: Ejemplo
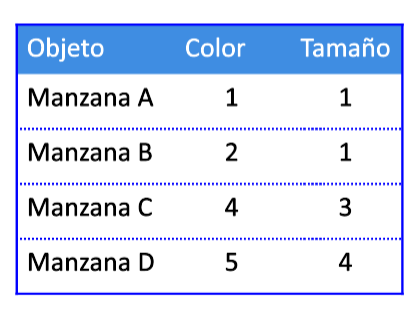
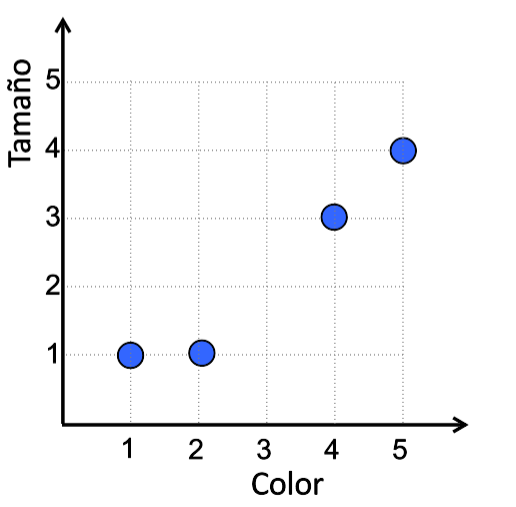
Resolvamos el siguiente ejemplo.
Supongamos que tenemos tipos de manzana, y cada una de ellas tiene 2 atributos (features). Agrupemos estos objetos en 2 grupos de manzanas basados en sus características.
K-Means: Ejemplo
1era Iteración
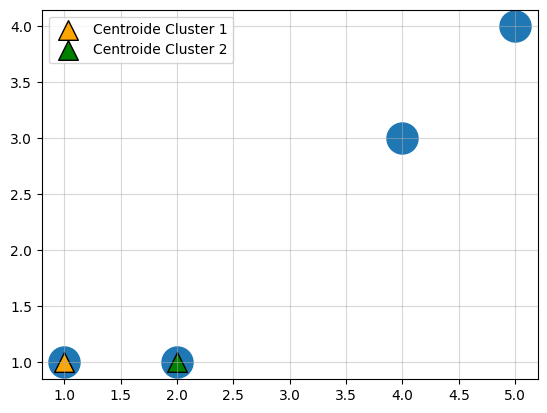
- Supongamos los siguientes centroides iniciales: \[C_1 = (1,1)\] \[C_2 = (2,1)\]
Matriz de Distancias al Centroide: (coordenada i,j representa distancia del punto j al centroide i)
\[D^1 = \begin{bmatrix} 0 & 1 & 3.61 & 5\\ 1 & 0 & 2.83 & 4.24 \end{bmatrix}\]
- Calculemos la Matriz de Pertenencia \(G\):
\[G^1 = \begin{bmatrix} 1 & 0 & 0 & 0 \\ 0 & 1 & 1 & 1 \end{bmatrix}\]
K-Means: Ejemplo
2da Iteración
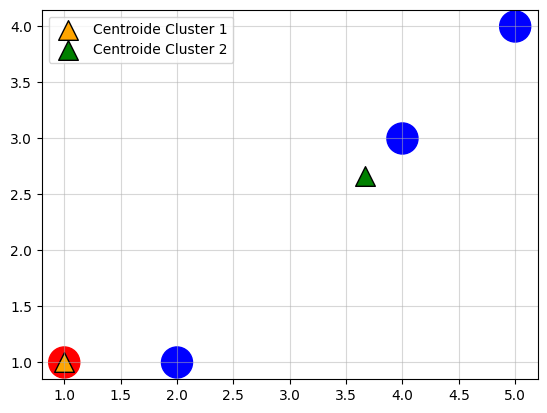
- Los nuevos centroides son:
\[C_1 = (1,1)\] \[C_2 = (\frac{11}{3}, \frac{8}{3})\]
- Calculamos la Matriz de Distancias al Centroide:
\[D^2 = \begin{bmatrix} 0 & 1 & 3.61 & 5\\ 3.14 & 2.26 & 0.47 & 1.89 \end{bmatrix}\]
- Calculemos la Matriz de Pertenencia \(G\):
\[G^2 = \begin{bmatrix} 1 & 1 & 0 & 0 \\ 0 & 0 & 1 & 1 \end{bmatrix}\]
K-Means: Ejemplo
- Si seguimos iterando notaremos que ya no hay cambios en los clusters. El algoritmo converge.
- Este es el resultado de usar \(K=2\). Utilizar otro valor de \(K\) entregará valores distintos.
- ¿Es este el número de clusters óptimos?
K-Means: Número de Clusters Óptimos
K-Means: Número de Clusters Óptimos
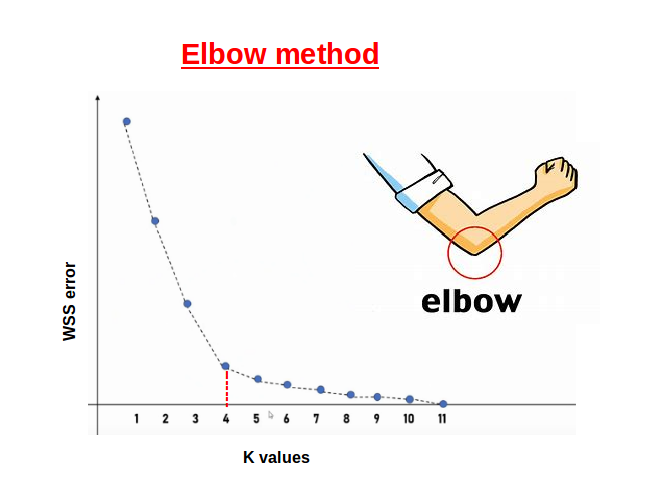
- Curva del Codo
- Es una heurísitca en la cual gráfica el valor de una métrica de distancia (e.g. within distance) para distintos valores de \(K\). El valor óptimo de \(K\) será el codo de la curva, que es el valor donde se estabiliza la métrica.
K-Means: Detalles Técnicos
Fortalezas
- Algoritmo relativamente eficiente (\(O(k \cdot n \cdot i)\)). Donde \(k\) es el número de clusters, \(n\) el número de puntos, e \(i\) el número de iteraciones.
- Encuentra “clusters esféricos”.
Debilidades
- Sensible al punto de inicio.
- Solo se puede aplicar cuando el promedio es calculable.
- Se requiere definir K a priori (K es un hiperparámetro).
- Suceptible al ruido y a mínimos locales (podría no converger).
Implementación en Scikit-Learn
from sklearn.cluster import KMeans
km = KMeans(n_clusters=8, n_init=10,random_state=None)
km.fit(X)
km.predict(X)
## opcionalmente
km.fit_predict(X)n_clusters: Define el número de clusters a crear, por defecto 8.
n_init: Cuántas veces se ejecuta el algoritmo, por defecto 10.
random_state: Define la semilla aleatoria. Por defecto sin semilla.
init: Permite agregar centroides de manera manual.
.fit(): Entrenará el modelo en los datos suministrados..predict()Entregará las clusters asignados a cada dato suministrado..clusters_centers_: Entregará las coordenadas de los centroides de cada Cluster..inertia_: Entrega valores correspondiente a lawithin cluster distance.
👀 Veamos un ejemplo en Colab.
Sugerencias
Pre-procesamientos
Es importante recordar que K-Means es un Algoritmo basado en distancias, por lo tanto se ve afectado por Outliers y por Escala.
Se recomienda preprocesar los datos con:
Winsorizer()para eliminar Outliers.StandardScaler()oMinMaxScaler()para llevar a una escala común.
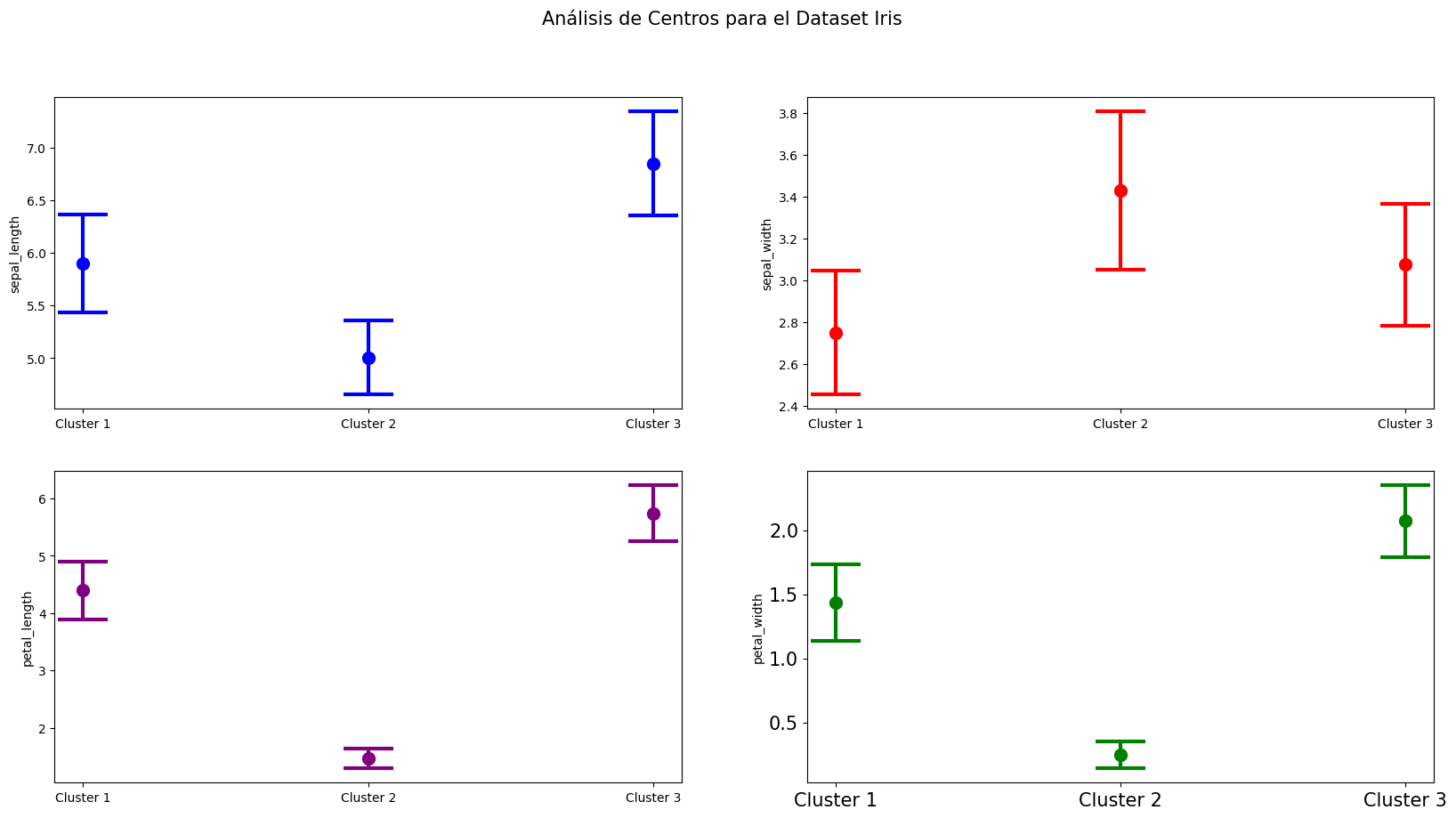
Interpretación Clusters
Recordar, que el clustering no clasifica. Por lo tanto, a pesar de que K-Means nos indica a qué cluster pertenece cierto punto, debemos interpretar cada cluster para entender qué es lo que se agrupó.
La interpretación del cluster es principalmente intuición y exploración, por lo tanto el EDA puede ser de utilidad para analizar clusters.
Post-Procesamiento: Merge
- Post-Procesamiento
-
Se define como el
tratamientoque podemos realizar al algoritmo luego de haber entregado ya sus predicciones.
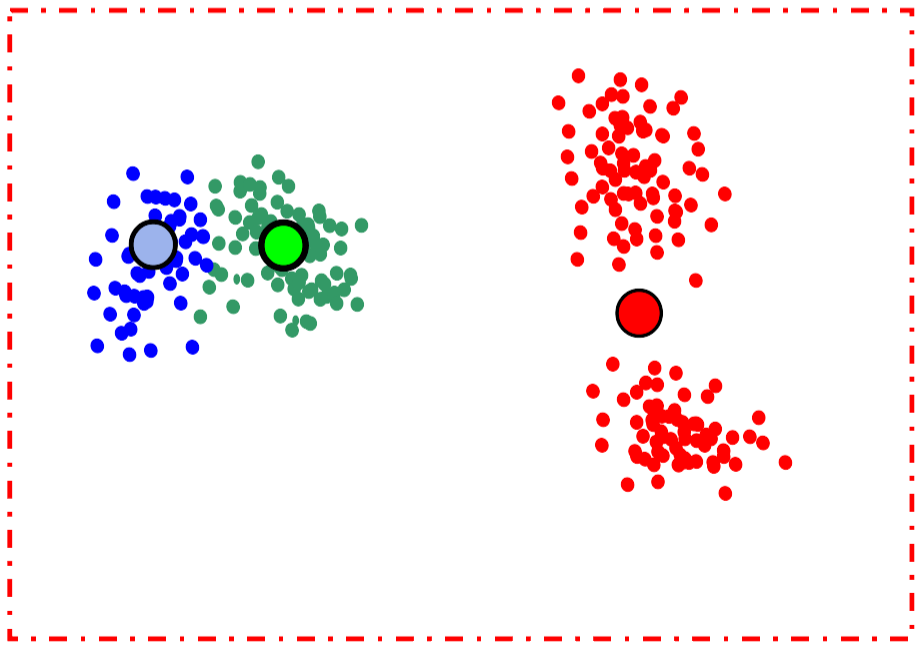
Es posible generar más clusters de los necesarios y luego ir agrupando los más cercanos.
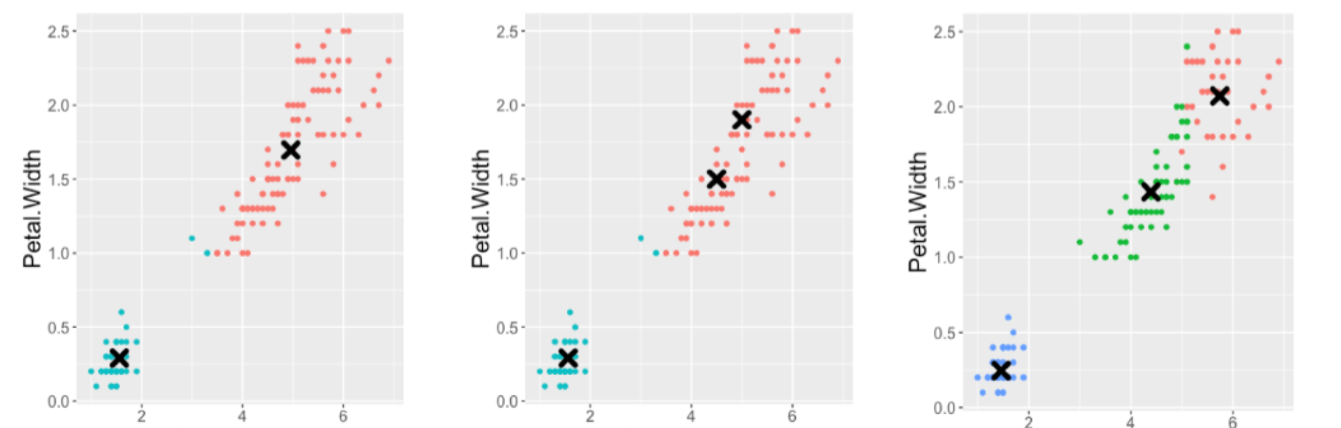
Post-Procesamiento: Merge
¿Cuál es el problema con este caso de Post-Procesamiento?
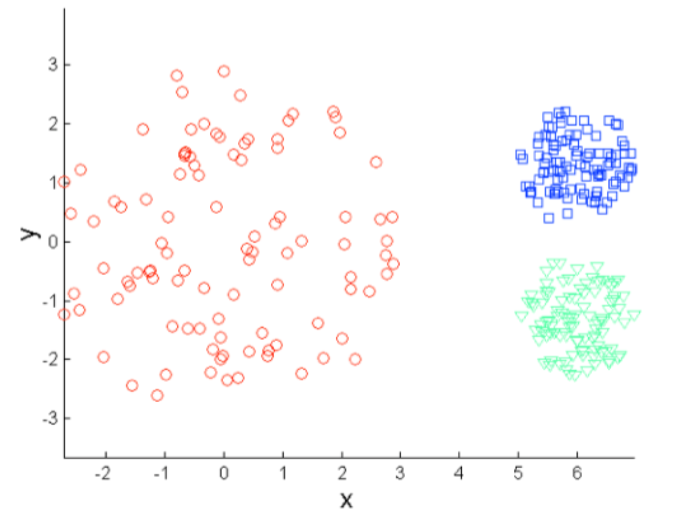
Post-Procesamiento: Split
En Scikit-Learn esto puede conseguirse utilizando el parámetro init. Se entregan los nuevos centroides para forzar a K-Means que separe ciertos clusters.
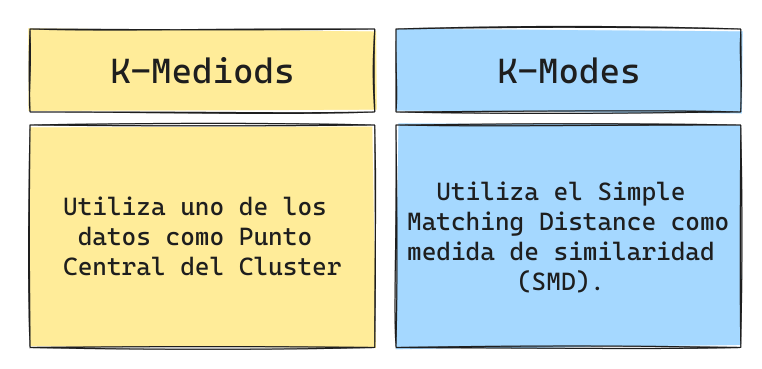
Variantes K-Means
Acá pueden encontrar una implementación de K-Modes en Python.
That’s all Folks