Fallando hasta el Éxito con Hydra
PyconChile 2021
Bueno esta semana me tocó presentar en la Pycon. Fue super buena la experiencia y a pesar de la presión propia de presentar de un tema que sea relevante para una audiencia que quiere aprender de Python me sentí super bien. Agradecer no más a Cristián Maureira que estuvo ahí ayudándome en la interna, super buena onda.
Obviamente una Pycon habla de todo, y no todo es el tema de interés de uno, pero si tuviera que destacar una charla sería la de Omar Sanseviero quien es ingeniero de HuggingFace. Omar mostró cómo hacer Demos de ML con Spaces, una nueva plataforma gratuita que lanzó HuggingFace para hostear aplicaciones en Gradio o Streamlit. Así que voy a estar chequeando cómo funciona y claramente voy a tratar de llevar mis modelos entrenados a un entorno productivo.
Bueno, el post de hoy es para compartir lo que hablé en mi Charla, Fallando hasta el Éxito con Hydra, en el cual explico cómo aprovechar este framework para poder entrenar distintas configuraciones de modelos de Machine Learning y conbinarlo con Optuna para la búsqueda de hiperparámetros. Hydra es una herramienta que ya utilicé en el pasado y si les interesa entender cómo funciona en otros contextos pueden ver mi antiguo post de Hydra acá.
La charla también está en Youtube y pueden toda la parte introductoria de la chala:
El post va a estar enfocado en el problema planteado durante mi presentación y el código utilizado para resolverlo. Además, explicaré algunos detallitos bien técnicos de la implementación presentada que no pude explicar debido a la duración de la charla (y también para no entrar en tecnicismos innecesarios que espantan a la audiencia).
El problema
Se quiere encontrar cuál es el modelo óptimo que resuelve un problema de clasificación binaria teniendo lo siguiente:
Set de Datos de Entrenamiento y Test en dos tamaños:
- Small (el que incluye sólo features numéricas)
- Large (que incluye también features categóricas)
Ambos Data Sets necesitan pasar por un preprocesamiento debido a que:
- Las variables numéricas y categóricas contienen NAs.
- Es necesario usar un Encoding en Variables Categóricas.
Para efectos de la demostración se quiere probar 2 modelos distintos para resolver el problema:
- Regresión Logística
- Random Forest
Una regresión Logística en Scikit-Learn
Para resolver este problema en Scikit-Learn se requiere un Script más o menos así:
import pandas as pd
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import accuracy_score
from feature_engine.imputation import MeanMedianImputer
Se hacen los imports necesarios para poder llevar a cabo el modelo:
- Importación de los Sets de Datos por medio de Pandas
- Modelo de Regresión Logística.
- Preprocesamientos usando Scikit-Learn y Feature-Engine (para imputar variables numéricas).
- Medición de la performance usando Accuracy.
train_df_small = pd.read_csv('data/data-train-small.csv')
test_df_small = pd.read_csv('data/data-test-small.csv')
X_train = train_df_small.drop(columns = 'Survived')
y_train = train_df_small.Survived
X_test, = test_df_small.drop(columns = 'Survived')
y_test = test_df_small.Survived
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method='mean')),
('sc', StandardScaler())
])
En este caso en particular estamos imputando valores perdidos con la media y Estandarizando (restando la media y dividiendo por desviación Estándar.)
model_pipe = Pipeline(steps = [
('prep', preprocess),
('model', LogisticRegression(random_state=123))
])
model_pipe.fit(X_train,y_train)
y_pred = model_pipe.predict(X_test)
print("El accuracy obtenido es:", accuracy_score(y_test, y_pred))
Y si quiero entrenar un Random Forest?
import pandas as pd
from sklearn.ensemble import RandomForestClassifier
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import accuracy_score
from feature_engine.imputation import MeanMedianImputer
train_df_small = pd.read_csv('data/data-train-small.csv')
test_df_small = pd.read_csv('data/data-test-small.csv')
X_train, y_train = train_df_small.drop(columns = 'Survived'), train_df_small.Survived
X_test, y_test = test_df_small.drop(columns = 'Survived'), test_df_small.Survived
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method='mean')),
('sc', StandardScaler())
])
model_pipe = Pipeline(steps = [
('prep', preprocess),
('model', RandomForestClassifier(
n_estimators = 100,
max_depth = 5,
min_samples_split = 10,
random_state=123))
])
model_pipe.fit(X_train,y_train)
y_pred = model_pipe.predict(X_test)
print("El accuracy obtenido es:", accuracy_score(y_test, y_pred))
La verdad es que el código es prácticamente igual, salvo que solo se debe importar la clase RandomForestClassifier y llamar el modelo al final del Pipeline de entrenamiento.
No sería mejor tratar de evitar la Duplicación?
Aquí es donde el principio de los males parte. Ya que como Data Scientist tenemos la tendencia de trabajar rápido y apurados no siguiendo siempre buenas prácticas. Las soluciones que yo he visto para esto son:
- Copiar el código en dos celdas de un Jupyter Notebook una con Random Forest (RF) y otra con Logistic Regression (LR).
- Comentar y descomentar el código del RF o de la LR dependiendo del que me interese ejecutar.
- Soluciones más
elegantes
tendrán un Script para el LR y otro para el RF.
Pero independiente de la solución, todas estas soluciones son poco prácticas y dificiles de mantener. Cualquier cambio que haya que hacer en el código significa tener que replicarlo en varios lugares a la vez lo que lo hace propenso a inconsistencias y bugs difíciles de rastrear.
Y en el caso que también quiera cambiar mi Dataset?
Por ejemplo en el caso que ahora quisiera entrenar el modelo utilizando el dataset large, tendría que modificar no sólo la importación de la data, sino que también el preprocesamiento:
train_df_small = pd.read_csv('data/data-train-large.csv')
test_df_small = pd.read_csv('data/data-test-large.csv')
X_train = train_df_small.drop(columns = 'Survived')
y_train = train_df_small.Survived
X_test, = test_df_small.drop(columns = 'Survived')
y_test = test_df_small.Survived
Esta parte es muy intuitiva y uno siempre nota la necesidad del cambio.
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method='mean')),
('imp_cat', CategoricalImputer(imputation_method='frequent')),
('ohe', OneHotEncoder()),
('sc', StandardScaler())
])
Pero la parte del preprocesamiento no es tan intuitiva, y la razón por la que es necesario cambiarla es porque ahora uno necesita generar imputación a variables categóricas (no consideradas en el dataset small) y además un encoding apropiado para que el modelo puede entender data no numérica.
El cambio en el código acá es mucho más invasivo y se empieza a entender la necesidad de poder ir rastreando todos los cambios que pueden depender de la data a utilizar, el preprocesamiento y el algoritmo + sus hiperparámetros.
La solución propuesta por Hydra
Hydrapropone utilizar archivos de configuración para poder ingresar todos los cambios del código desde un archivo de manera ordenada.- Además
Hydragenera una carpeta con la fecha y hora con los outputs y la configuración utilizada de cada ejecución. De esa manera es posible llevar registro de manera mucho más ordenada de los experimentos realizados y sus resultados y de los que falta por hacer.
Una cosa que no mencioné en la presentación es que Hydra posee Callbacks. Esto permitiría, por ejemplo, ir logueando el resultado de los experimentos en plataformas como Weights & Biases (se viene también un tutorial de cómo utilizarlo pronto).
Refactorizando el código utilizando Hydra obtenemos lo siguiente:
from omegaconf import DictConfig
import hydra
from hydra.utils import to_absolute_path
import pandas as pd
from preprocessing import simple_preprocess, complex_preprocess
from sklearn.linear_model import LogisticRegression
from sklearn.ensemble import RandomForestClassifier
from sklearn.pipeline import Pipeline
from sklearn.metrics import accuracy_score
En este caso, debemos importar Hydra, DictConfig de omegaconf (una depencia que se instala automaticamente con Hydra), y to_abolute_path.
@hydra.main(config_path ='conf', config_name = 'config')
def train_model(cfg: DictConfig):
train_df_small = pd.read_csv(to_absolute_path(cfg.preprocess.data_train))
test_df_small = pd.read_csv(to_absolute_path(cfg.preprocess.data_test))
X_train, y_train = train_df_small.drop(columns = 'Survived'), train_df_small.Survived
X_test, y_test = test_df_small.drop(columns = 'Survived'), test_df_small.Survived
preprocess = hydra.utils.call(cfg.preprocess.type)
model_pipe = Pipeline(steps = [
('prep', preprocess),
('model', hydra.utils.instantiate(cfg.models.type))
])
model_pipe.fit(X_train,y_train)
y_pred = model_pipe.predict(X_test)
print(f"El Accuracy obtenido por {cfg.models.name} es:", accuracy_score(y_test, y_pred))
- Como hemos visto en tutoriales anteriores, todo el código de
Hydradebe ser envuelto en una función decorada con@hydra.mainel cual indicará la ubicación de los archivos de configuración. - La función debe tener un único parámetro de configuración anotado como
DictConfiglo que permitirá extraer los parámetros de los archivos de configuración. - Los valores antepuestos de
cfg.___permiten recatar los parámetros de configuración del archivo que corresponda. hydra.utils.callpermitirá invocar funciones, que en nuestro caso serán las funciones de preprocesamiento dependiendo del dataset a utilizar.hydra.utils.instantiatepermitirá instanciar Clases (en nuestro caso deScikit-Learn) que representan los modelos que queremos entrenar (LogisticRegressionoRandomForestClassifier).
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from feature_engine.imputation import CategoricalImputer, MeanMedianImputer
from feature_engine.encoding import OneHotEncoder
def simple_preprocess(imputation_method):
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method=imputation_method)),
('sc', StandardScaler())
])
return preprocess
def complex_preprocess(imputation_num, imputation_cat):
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method=imputation_num)),
('imp_cat', CategoricalImputer(imputation_method=imputation_cat)),
('ohe', OneHotEncoder()),
('sc', StandardScaler())
])
return preprocess
Este Script que está siendo importado en el Script principal del proyecto y contiene dos funciones las cuales son las encargadas de aplicar un preprocesamiento adecuado dependiendo del tipo de data.
simple_preprocesscontiene sólo imputación con la media para variables numéricas y estandarización.complex_preprocessagrega imputación categórica por medio de la moda yOneHotEncoder.
Finalmente los archivos de configuración quedan como siguen:
type:
_target_: main.simple_preprocess
imputation_method: mean
data_train: data/data-train-small.csv
data_test: data/data-test-small.csv
type:
_target_: main.complex_preprocess
imputation_num: mean
imputation_cat: frequent
data_train: data/data-train-large.csv
data_test: data/data-test-large.csv
name: Logistic Regression
type:
_target_: main.LogisticRegression
C: 1
random_state: 123
name: Random Forest
type:
_target_: main.RandomForestClassifier
n_estimators: 100
max_depth: 5
min_samples_split: 10
n_jobs: -1
random_state: 123
Esto se logra utilizando la siguiente estructura de Carpetas:
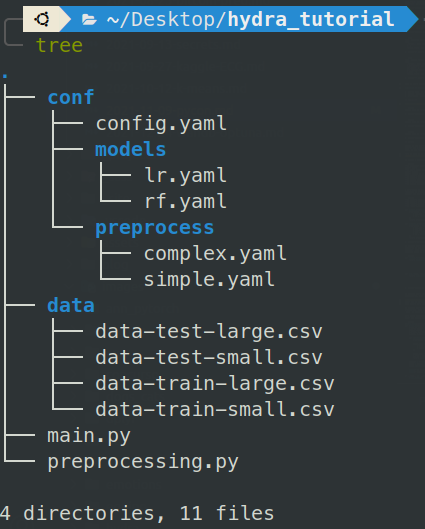
config.yamles nuestra configuración global, la cual por ahora no tiene nadamodelscontiene la configuración de los modelos a intanciar.preprocesscontiene el preprocesamiento a utilizar y la data compatible para dicho preprocesamiento.
Ejecución
Finalmente Hydra, automáticamente crea una línea de comando que permite ir probando las configuraciones implementadas.
python main.py +preprocess=simple +models=lr
python main.py +preprocess=complex +models=rf
python main.py +preprocess=complex +models=lr ++models/C=0.1
Optuna
Optuna es una librería de Optimización Bayesiana que es de las más top en Kaggle. Su implementación es sencilla y permite generar una búsqueda de hiperparámetros basada en resultados del pasado. Este es un framework que requiere de un tutorial por sí sola, ya que realmente tiene muchas opciones.
Para el caso de la charla, sólo mostré como maximizar una función objetivo (que resulta ser el modelo a entrenar) y cómo Hydra permite crear muchos modelos rápidamente:
El código a implementar es el siguiente:
from omegaconf import DictConfig
import hydra
from hydra.utils import to_absolute_path
import optuna
import pandas as pd
from sklearn.pipeline import Pipeline
from sklearn.metrics import accuracy_score
from models import random_forest, log_reg
from preprocessing_multirun import simple_preprocess, complex_preprocess
from optuna.samplers import TPESampler
A diferencia de los casos anteriores hice un nuevo script llamadado preprocessing_multirun.py en el cual cada una de las funciones de prepocesamiento las modifiqué para que permitan el parámetro trial requerido por Optuna. Además los valores a imputar son ahora sugerencias que serán sampleados utilizando la metodología Bayesiana implementada por Optuna.
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from feature_engine.imputation import CategoricalImputer, MeanMedianImputer
from feature_engine.encoding import OneHotEncoder
def simple_preprocess(imp_method_values, trial=None):
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method=trial.suggest_categorical("imp_num", imp_method_values))),
('sc', StandardScaler())
])
return preprocess
def complex_preprocess(imputation_num, imputation_cat, trial=None):
preprocess = Pipeline(steps = [
('imp_num', MeanMedianImputer(imputation_method=trial.suggest_categorical("imp_num", imputation_num))),
('imp_cat', CategoricalImputer(imputation_method=trial.suggest_categorical("imp_cat", imputation_cat))),
('ohe', OneHotEncoder()),
('sc', StandardScaler())
])
return preprocess
Acá hice la pillería de utilizar trial como un parámetro opcional con valor por defecto None. Esto es necesario ya que trial es la manera en la que Optuna indica que está tomando una sugerencia dependiendo del sampleo Bayesiano a utilizar. Como estas funciones están definidas en otro módulo de Python, trial no está definido. Dejándolo como parámetro opcional evito el error.
def random_forest(n_estimators, max_depth, min_samples_split, trial = None):
return RandomForestClassifier(n_estimators = trial.suggest_int('n_estimators', **n_estimators),
max_depth = trial.suggest_int('max_depth', **max_depth),
min_samples_split = trial.suggest_discrete_uniform('min_samples_split', **min_samples_split),
n_jobs = -1,
random_state = 123
)
def log_reg(C, fit_intercept, trial = None):
return LogisticRegression(C = trial.suggest_loguniform('C', **C),
fit_intercept=trial.suggest_categorical('fit_intercept', fit_intercept),
n_jobs = -1,
random_state = 123
)
Si se fijan tuve que hacer una pillería similar para los modelos, debido a que los modelos deben instanciarse, sólo pueden instanciarse de manera “parámetrica” dentro de una función, por eso esta transformación.
Luego la función decorada de Hydra contendrá en su interior optimize_model() la cual será la función a Optimizar. Se podría decir que la definición de una función dentro de una función no es lo más correcto, pero en este caso tenemos que hacerlo de esta manera para poder aprovechar los parámetros provenientes de los archivos de configuración.
Es sumamente importante que los llamados de preprocesamiento y Modelos contengan el kwarg trial = trial. Esto, para que trial deje de ser None que es su valor por defecto. De no hacerlo, obtendremos como error que un None no tiene el método suggest_*.
Otro punto sumamente importante que recalco en la charla es que la función a optimizar debe tener un return, que es es el valor que Optuna intenta maximizar. En nuestro caso queremos obtener el mejor Accuracy posible.
@hydra.main(config_name='config', config_path='conf')
def train_model(cfg: DictConfig):
train_df_small = pd.read_csv(to_absolute_path(cfg.preprocess.data_train))
test_df_small = pd.read_csv(to_absolute_path(cfg.preprocess.data_test))
X_train, y_train = train_df_small.drop(columns = 'Survived'), train_df_small.Survived
X_test, y_test = test_df_small.drop(columns = 'Survived'), test_df_small.Survived
def optimize_model(trial):
preprocess = hydra.utils.call(cfg.preprocess.type, trial = trial)
model_pipe = Pipeline(steps = [
('prep', preprocess),
('model', hydra.utils.call(cfg.models.type, trial = trial))
])
model_pipe.fit(X_train,y_train)
y_pred = model_pipe.predict(X_test)
return accuracy_score(y_test, y_pred)
sampler = TPESampler(seed=123)
study = optuna.create_study(sampler = sampler, direction="maximize")
study.optimize(optimize_model, n_trials=cfg.n_trials)
print(f'El mejor accuracy conseguido fue: {study.best_value}')
print(f'usando los siguientes parámetros: \n \t \t{study.best_params}')
if __name__ == '__main__':
train_model()
Finalmente es importante recalcar que el muestreo Bayesiano es reproducible y que es necesario indicar el número de ensayos (muestras) a generar. Esto equivale en nuestro caso a el número de modelos a entrenar.
Notar que los Archivos de Configuración definirán un rango de Hiperparámetros que serán los espacios de búsqueda que Optuna utilizará para entrenar los modelos solicitados. Los archivos de configuración quedan de la siguiente manera:
name: Multi Logistic Regression
type:
_target_: main.log_reg
C:
low: 0.001
high: 100
fit_intercept: [false, true]
name: Multi Random Forest
type:
_target_: main.random_forest
n_estimators:
low: 10
high: 600
step: 10
max_depth:
low: 1
high: 10
step: 1
min_samples_split:
low: 0.1
high: 1
q: 0.1
–
type:
_target_: main.simple_preprocess
imp_method_values: [mean, median]
data_train: data/data-train-small.csv
data_test: data/data-test-small.csv
type:
_target_: main-multirun.complex_preprocess
imputation_num: [mean, median]
imputation_cat: [frequent, missing]
data_train: data/data-train-large.csv
data_test: data/data-test-large.csv
n_trials: ???
Como he mencionado en otros tutoriales, ??? permite definir un valor no definido previamente pero mandatorio. Por lo tanto, el código fallará a menos que se entregue este valor (que es el número de modelos) al momento de la ejecución.
Normalmente en una situación uno va a ir directamente a esta implementación. Y generará muchos archivos de configuración dependiendo de que tan exhaustiva quiere que sea la búsqueda. Mi recomendación es pasar mucho tiempo tratando de generar este código, permitiendo que sea lo más flexible posible y, cuando uno note que no falla, ejecutar todos los experimentos con el multirun. Dejas tu compu corriendo en la noche, y revisas todos los resultados en la mañana.
Ejecución Multirun
La ejecución multirun permitirá ejecutar todas las combinaciones diseñadas. Obviamente esto se puede volver tedioso en caso de tener 100 archivos de configuración. La manera en la que funciona el multirun es con el flag -m.
python main.py -m +preprocess=simple,complex +models=lr,rf +n_trials=100
En este caso ejecutaremos 100 modelos para las 4 combinaciones posible entre modelos y preprocesamientos.
Otra cosa que no mencioné en la charla es que para el multirun se soporta sintaxis tipo glob. Por lo tanto no es necesario listar todos los archivos de configuración en caso de que quieras ejecutar TODO LO EXISTENTE. Más info de esto acá.
Bueno, espero que este tutorial haya sido útil. Vean igual la charla, creo que a veces es más dinámico de entender y además en el final de la charla dejé un desafío. Disponibilicé todo un código más datos de manera funcional en este Repo para que puedan jugar a voluntad.
Espero les guste, y nos vemos a la otra.
